Estudio asegura que peligrosa variante británica se estaba expandiendo silenciosamente por el mundo mucho antes de ser descubierta
La investigación señala que la variante, descubierta en diciembre de 2020, ya se había expandido al menos a 15 países mucho antes de su hallazgo. Varios especialistas estiman que la letal cepa podría haber llegado a Chile en noviembre.

La llamada variante británica, altamente contagiosa del Sars-CoV-2 se propagó sin saberlo durante meses en varias partes del mundo mucho antes de ser detectada, según un nuevo estudio de investigadores del Consorcio de Modelado Covid-19 de la Universidad de Texas en Austin (EE.UU).
La variante, más contagiosa y letal de las que actualmente circulan, fue descubierta por primera vez a principios de diciembre en el Reino Unido, donde se cree que se originó. La revista Emerging Infectious Diseases, que publicó una versión preliminar del estudio, proporciona evidencia de que la variante del coronavirus, cuyo nombre científico es B117 (501Y), se había extendido por todo el mundo sin ser detectada varios meses antes de que los científicos la descubrieran.
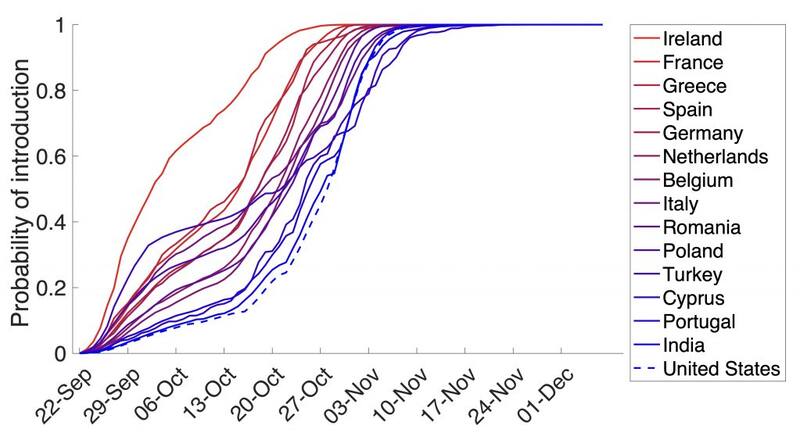
Los investigadores descubrieron que la variante apareció por primera vez en el Reino Unido a principios de diciembre de 2020, y luego se extendió a los EE. UU., Irlanda, Francia, Grecia, España, Alemania, los Países Bajos, Bélgica, Italia, Rumania, Polonia, Turquía, Chipre, Portugal e India.
“Cuando nos enteramos de la variante del Reino Unido en diciembre, ya se estaba extendiendo silenciosamente por todo el mundo”, dijo en un comunicado Lauren Ancel Meyers, directora del Consorcio de Modelado Covid-19 y profesora de biología integrativa. “Estimamos que la variante B117 probablemente llegó a EE.UU. en octubre de 2020, dos meses antes de que supiéramos que existía”.
Al analizar datos de 15 países, según la información proporcionada por la U. de Texas, los investigadores estimaron la posibilidad de que viajeros del Reino Unido introdujeran la variante en 15 países entre el 22 de septiembre y el 7 de diciembre de 2020. Descubrieron que la variante del virus casi con certeza había llegado a los 15 países a mediados de noviembre. En Estados Unidos, la variante probablemente llegó a mediados de octubre.
“Este estudio destaca la importancia de la vigilancia de laboratorio”, dijo Meyers. “La secuenciación rápida y extensa de muestras de virus es fundamental para la detección temprana y el seguimiento de nuevas variantes de interés”.
Junto con la publicación del artículo, los miembros del consorcio desarrollaron una nueva herramienta que los responsables de la toma de decisiones en cualquier lugar de los Estados Unidos pueden utilizar para planificar la secuenciación genética que ayuda a detectar la presencia de variantes.
Para ayudar a EE.UU. a expandir la vigilancia nacional de variantes, la nueva calculadora en línea indica la cantidad de muestras de virus que deben secuenciarse para detectar nuevas variantes cuando surgen por primera vez. Por ejemplo, si el objetivo es detectar una variante emergente en el momento en que está causando 1 de cada 1.000 nuevas infecciones por Covid-19, es necesario secuenciar aproximadamente 3.000 muestras positivas para el Sars-CoV-2 por semana.
“Los funcionarios de salud están buscando mejores formas de manejar la imprevisibilidad de este virus y las variantes futuras”, dijo Spencer Woody, becario postdoctoral en el Consorcio de Modelado UT Covid-19. “Nuestra nueva calculadora determina cuántas muestras positivas de Sars-CoV-2 deben secuenciarse para garantizar que se identifiquen nuevas amenazas tan pronto como comiencen a propagarse”, agregó.
Explicó que la calculadora tiene una segunda función. “También ayuda a los laboratorios a determinar la rapidez con la que detectarán nuevas variantes, dada su capacidad de secuenciación actual”.
“Creamos esta herramienta para apoyar a los funcionarios de salud federales, estatales y locales en la construcción de sistemas de alerta temprana creíbles para esta y futuras amenazas de pandemia”, dijo Meyers.
La investigación fue financiada por el Fondo de Investigación Médica y de Salud de Hong Kong, los Institutos Nacionales de Salud y los Centros para el Control y la Prevención de Enfermedades.
Variante británica podría haber llegado en noviembre a Chile
Según el último informe de circulación de variantes publicado este lunes por el Ministerio de Salud, al 26 de marzo en Chile se han identificado 64 casos de la variante británica B.1.1.7. De ellos 37 corresponden a viajeros, 18 a casos comunitarios y 9 a casos secundarios. El 55% de estos casos han sido sintomáticos y el 9% requirió hospitalización.
De la variante brasileña, conocida como P.1, se han encontrado 45: 34 corresponden a viajeros, 8 casos comunitarios y 3 a casos secundarios. De ellos, el 71% presentó síntomas y 4 casos requirieron hospitalización.
La infectóloga del Hospital Clínico de la Universidad de Chile, Jeannette Dabanch, señaló en una nota publicada por Qué Pasa, que la variante británica está circulando en el país, probablemente desde fines de noviembre, aunque se detectó por primera vez en diciembre del año pasado. Lo mismo ocurre con la variante brasileña.
Para poder saber qué variante está circulando en el país y por cuáles hay que preocuparse, el Ministerio de Ciencia, el Ministerio de Salud, el Instituto de Salud Pública (ISP) y algunas universidades, vienen trabajando en un Programa de Vigilancia Genómica. La idea es poder secuenciar más muestras de las que ahora secuencia el ISP y poder disponibilizar todos esos datos en el sistema que ya maneja el Ministerio de Ciencia.
A la fecha, ya llevan al menos tres sesiones de trabajo para coordinar todo este trabajo y se hizo un catastro único de capacidades nacionales (Consorcio de Universidades y la Sociedad Chilena de Genética) y un documento que establece estándares para la toma y procesamiento de muestras (liderado por la UC y la Universidad de Magallanes).
En este mismo reportaje, el director (s) del Instituto de Salud Pública, Heriberto García Escorza, señala que durante la vigilancia activa de Sars-CoV-2 en diferentes regiones del país, en los meses de enero, febrero y hasta el 11 de marzo del 2021, el Instituto de Salud Pública ha obtenido el genoma completo en 258 muestras” y solo durante los primeros 11 días de marzo, en 45 genomas completos obtenidos de muestras recolectadas en la Región Metropolitana, se identificaron 11 linajes diferentes: se detectaron los linajes de preocupación B.1.1.7 en 7 casos y P.1 en 5 casos.
García señaló sin embargo, que estas nuevas variantes no son las que predominan en el país. “Nosotros hemos logrado controlar la variante, la velocidad de aumento no es la que ocurrió en los otros países, avanzó más lento. Ni la variante británica ni la brasileña son las predominantes, están dentro de las que estamos estudiando, pero la variante N4 sigue siendo predominante desde hace varios meses y es la que llegó desde Europa”, dijo el director.
COMENTARIOS
Para comentar este artículo debes ser suscriptor.
Lo Último
Lo más leído
1.
3.



















